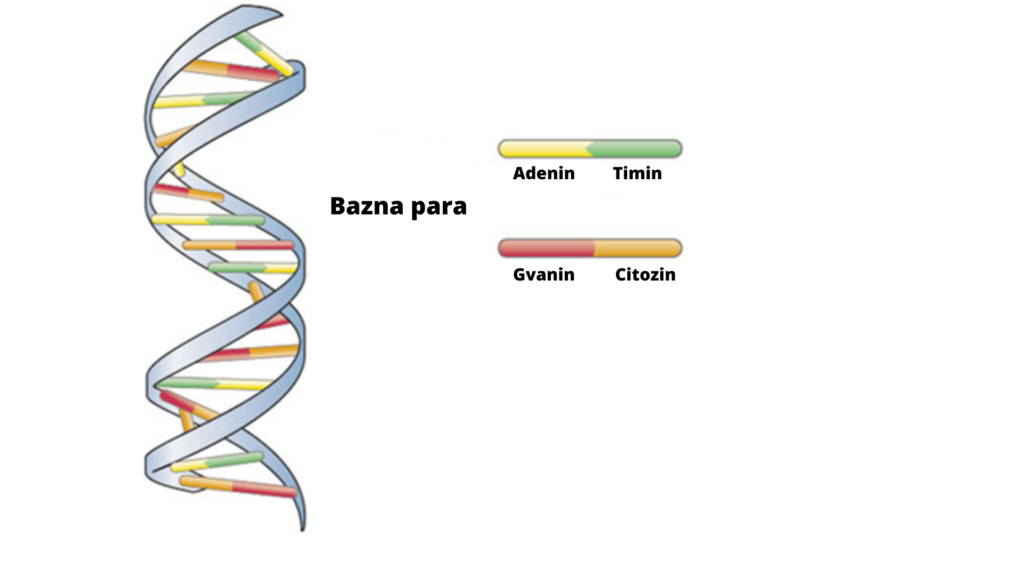
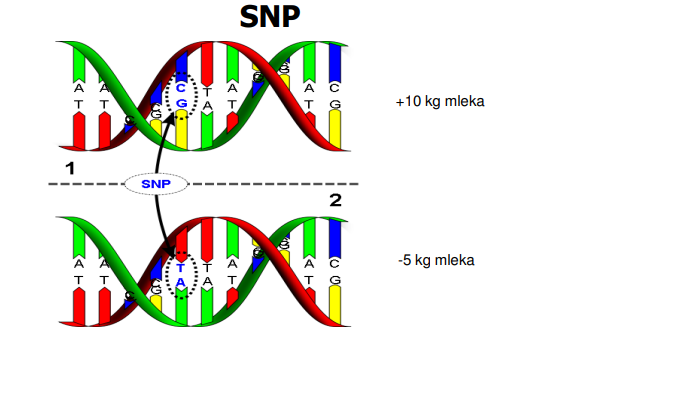
Polimorfizem posameznega nukleotida (ang. Single-Nucleotide Polymorphism SNP (izgovorjeno »SNIP«), je ena najpogostejših vzrokov genetske variacije med osebki znotraj vrste. Vsak SNP predstavlja spremembo v enem samem osnovnem gradniku DNA, ki se imenuje nukleotid (slika 1). Te spremembe so posledice mutacij, ki nastanejo pri podvajanju ali popravljanju DNA. Na sliki 2 je prikazan primer mutacije nukleotida citozin (C) v nukleotid timin (T), kar pomeni, da je to mesto polimorfno (= SNP). Sprememba v posameznem nukleotidu lahko povzroči spremembo v zaporedju aminokilsin oziroma strukturo proteina. Mutiran alel (genska varianta), ki ni škodljiv ali je celo ugoden za preživetveno sposobnost osebka, se skozi generacije razširi po populaciji. Posledično del populacije nosi mutiran alel, preostali del populacije pa prvoten divji tip alela (ang. Wild type allele«).
SNP-i so v DNA normalen pojav. V povprečju se SNP pojavi vsake 1.000 nukleotidov, kar pomeni, da v genomu nosimo okoli 4 do 5 milijonov SNP-jev. Te variacije se v zapisu posameznih odsekov genoma pojavljajo pri velikem številu osebkov v populaciji. Da se varianta opredeli kot SNP, se mora pojaviti pri najmanj 1% populacije.
SNP-i se najpogosteje pojavljajo v nekodirajočih regijah med samimi geni. Delujejo lahko kot biološki markerji, s katerimi si raziskovalci pomagajo pri iskanju genov, ki so povezani z lastnostjo ali obolenjem. SNP-i znotraj gena ali regulatorne regije blizu gena lahko vplivajo na delovanje ali izražanje gena in posledično na fenotip. Večina SNP-ov nima vpliva na razvoj ali zdravje in jih uporabljamo kot genetske označevalce oz. markerje. Vseeno so ti SNP-i zelo pomembni za raziskovalne namene, saj jih lahko uporabljamo za napovedi genomskih plemenskih vrednosti. Nevzročni SNP-i so povezani z vzročnimi geni in nam omogočajo napoved proizvodnih sposobnosti živali, tveganja za obolenje ter odziva na dano okolje. SNP-i predstavljajo unikaten prstni odtis posameznika in se lahko uporabi za testiranje porekla.
Celotno sekvenco genoma govedi so objavili v letu 2009. Goveji genom vsebuje 29 parov avtosomalnih kromosomov in en par spolnih kromosomov, skupno 60. Genom vsebuje skupno 3 miliarde baznih parov in približno 22.000 genov, od katerih je 80% homolognih s človekom (homologni gen, DNA zaporedje ali protein je definiran v smislu skupnega porekla, homologna segmenta DNA dveh različnih vrst imata skupnega prednika ).
Vezavno neravnovesje
Rekombinacija je proces, ki omogoča nastanek genetsko raznolikih spolnih celic. Pri tem procesu se s prekrižanjem homolognih kromosomov in gensko konverzijo izmenjajo deli DNA med dvema DNA vijačnicama. Ali se bosta dva SNP-a po rekombinaciji še vedno nahajala na isti molekuli DNA, je odvisno od njune medsebojne razdalje. Kadar sta locirana v bližini drug drugega, je zelo verjetno, da do rekombinacije med njima ne bo prišlo in se bosta dedovala skupaj. Temu pravimo, da sta v vezavnem neravnovesju. Alelne frekvence SNP-ov se deloma razlikujejo med različnimi populacijami in so pasemsko značilne. Razlike med populacijam povzročajo preplet rekombinacije, naključnega genetskega toka in ločene evolucijske poti populacij. Rezultati genotipizacije ene populacije ne predstavljajo druge.
Genomska selekcija
Genomska selekcija izbira osebke na podlagi plemenskih vrednosti, ocenjenih na osnovi genomskih podatkov. SNP-e uporablja kot genetske markerje, ki služijo kot markerji genov in njihovih alelov. Da se lahko SNP uporabi kot genski marker, mora ležati na istem lokusu, mestu na kromosomu, na katerem je gen. SNP-i, ki jih uporabimo za napoved genomskih plemenskih vrednosti ( = markerji na SNP čipu), so v vezavnem neravnovesju z lokusi, ki vplivajo na kvantitativne lastnosti (QTL) . Ta pristop je postal mogoč šele z odkritjem večjega števila SNP-ov, odkritih s pomočjo sekvenciranja (branja) genoma.
Genotipiziranje s SNP čipi
Pri SNP genotipiziranju ugotavljamo, katero varianto alela nosi posameznik. Porast zanimanja za SNP genotipiziranje, se odraža v skokovitem razvoju raznolikih SNP metod in tehnologij za genotipiziranje.
Kako deluje SNP čip?
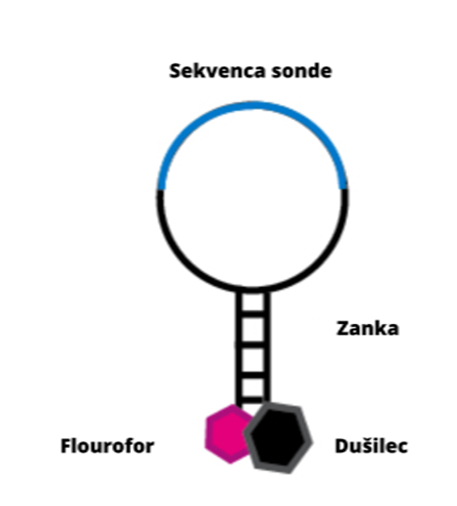
Za določanje alela na SNP-ih se na čipih za genotipiziranje uporabljajo posebej razvite enoverižne oligonukleotidne sonde, ki imajo na vsakem koncu komplementarni regiji ter tarčno zaporednje nukleotidov v srednjem delu (slika 3). Oligonutkleotid je krajša veriga DNA ali RNK, dolga do 20 nukleotidov. Takšna struktura omogoča sondi, da v svojem naravnem izoliranem stanju zavzame strukturo zanke. Na enem koncu sonde je pritrjen fluorofor (molekula, ki oddaja fluorescentno svetlobo), na drugem koncu pa dušilec fluorescence. V naravnem stanju je tako fluorofor v neposredni bližini dušilca, kar preprečuje, da bi molekula fluorophora oddala fluorescentno svetlobo. Sonda je zasnovana tako, da je sekvenca nukleotidov v sredi sonde komplementarna genomski DNA, ki bo uporabljena v testu.
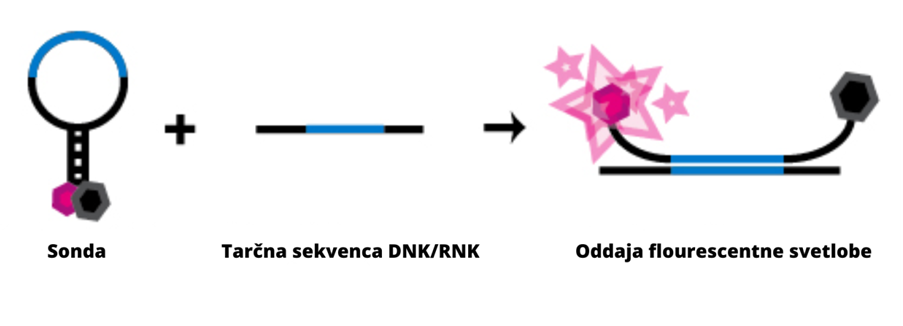
Ko se sonda na čipu sreča s tarčno DNA, se z njo hibridizira, kar povzroči, da se razpre. Sprememba oblike omogoča, da fluorofor prične oddajati fluorescentno svetlobo (slika 4).
Že en sam nepravilen nukleotid (SNP) v tarčni DNA je dovolj, da sonda ne prime in ostane v svoji prvotni obliki zanke. V tem primeru na čipu ni mogoče zaznati flourescentne svetlobe, saj fluorofor in dušilec ostaneta v neposredni bližini drug drugega.
Zgradba sonde omogoča preprosto identifikacijo SNP-jev na izbrani lokaciji (slika 5). Če je ena sonda na čipu komplementarna divjemu (nemutiranemu) tipu alela, druga sonda pa mutiranemu tipu alela, lahko ti dve uporabimo za identifikacijo genotipa posameznika. Če naprava, ki zaznava valovne dolžine fluorescentne svetlobe, prepozna le svetlobo prve sonde, je posameznik »wild type« homozigot (ima dva nemutirana alela). Če naprava prepozna le svetlobo druge sonde, je osebek homozigot z dvema mutiranima aleloma. Če naprava prepozna obe valovni dolžini fluorescentne svetlobe, pomeni, da sta se hibridizirali obe sondi in je osebek heterozigot (ima genotip z mutiranim in nemutiranim alelom).
Tehnologija nanopor
Najnovejša generacija naprav za sekvenciranje DNA uporablja nekoliko drugačen pristop za branje zaporedja DNA. Naprave imajo vgrajene pretočne celice, na katerih so nameščeni proteini nanopor, ki hkrati prepuščajo in sekvencirajo DNA. Ob prehodu DNA skozi nanoporo se v membrani pretočne celice ustvarja šibek električni tok. Vsak od štirih baznih parov povzroči edinstven signal, katerega programska oprema simultano beleži kot zaporedje nukleotidov (slika 6).
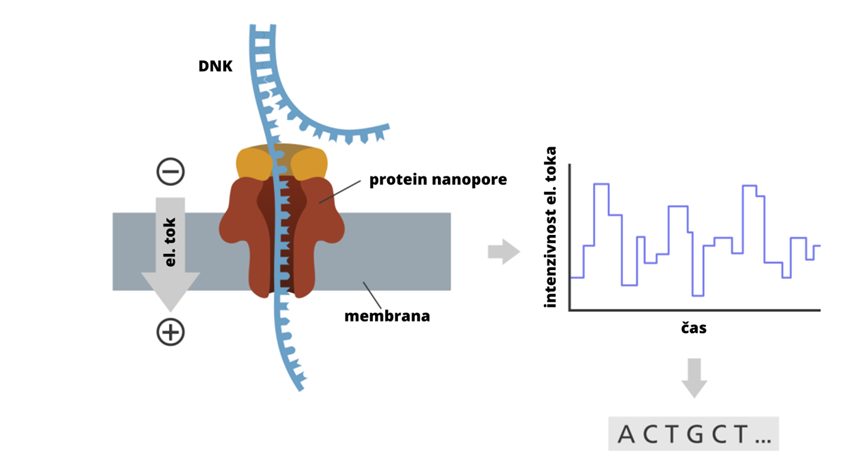
Prednost nanopor tehnologije je, da nima omejitev glede dolžine odseka DNA, olajša sekvenciranje težkih zaporedij ponavljajočih se regij DNA brez potrebe po naknadni obdelavi DNA ter razrešuje velike strukturne variacije in med sabo razlikuje izoforme. Omejene so le z dolžino DNA, ki prehaja skozi poro. Za genotipizacijo ni več potrebno pomnoževanje DNA/RNK s polimerazo, poleg prepoznavanja sekvence nukleotidov pa je omogočeno tudi prepoznavanje epigenetskih modifikacij baznih parov, kot je na primer metilacija.
Avtor: Urban Vodopivc, KIS
VIRI:
Elsik C. G. Tellam R. L, Worley K. C. 2009. Bovine Genome Sequencing and Analysis Consortium. The genome sequence of taurine cattle: a window to ruminant biology and evolution. Science, 324, 5926: 522–528
Goddard M. E. in Hayes B. J. 2007. Genomic selection. Journal of Animal Breeding and Genetics, 124, 6: 323-330
Wang Y., Zhao Y., Bollas A., Wang Y., Au K. F. 2021. Nanopore sequencing technology, bioinformatics and applications. Nature Biotechnology, 39: 1348–1365